El reposicionamiento de fármacos es una técnica innovadora en la investigación farmacéutica que consiste en dar nuevos usos a medicamentos ya existentes. Una gran ventaja de esta práctica es poder utilizar fármacos para enfermedades distintas a las que generalmente son aplicadas, reduciendo significativamente el coste y riesgo asociado a la creación de nuevos medicamentos desde cero.
Según investigaciones del CSIC, el 75% de los fármacos conocidos pueden tener nuevos usos terapéuticos. Además, se ha comprobado que descubrir nuevas interacciones entre medicamentos y proteínas ayuda al proceso de desarrollo de fármacos, reduciendo considerablemente el tiempo y coste del proceso. Por tanto, en los últimos años se ha convertido en una vía prometedora en el mundo farmacéutico, e incluso desde el punto de vista del aprendizaje automático o Machine Learning. En este artículo, investigamos cómo estas técnicas de inteligencia artificial pueden ayudar a encontrar estos potenciales nuevos links.
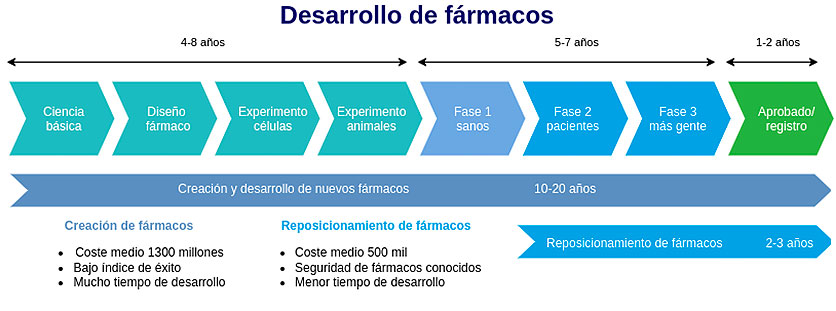
¿Cómo abordar el reposicionamiento de fármacos?
Este proceso de reposicionamiento de fármacos puede hacerse desde dos enfoques: uno más biológico, orientado a la estructura química de fármacos y las secuencias de proteínas y otro más algorítmico, orientado a las enfermedades relacionadas con los fármacos y las proteínas. La información necesaria de partida será diferente dependiendo del enfoque:
- Estructura química de los fármacos.
- Secuencia de aminoácidos de proteínas.
- Relaciones fármaco-enfermedad.
- Relaciones proteína-enfermedad.
- Interacciones fármaco-proteína.
En nuestro caso, vamos a predecir este último punto, centrándonos en analizar las enfermedades relacionadas con los fármacos y las proteínas, el enfoque más algorítmico.
Matrices de similitud
Los elementos más importantes de este estudio son la matriz de similitud entre los fármacos y la matriz de similitud entre las proteínas. Estas matrices proporcionan una intuición de cuánto se parecen cada par de fármacos entre sí y cada par de proteínas entre sí. Dependiendo del enfoque elegido, el parecido será en base a su estructura interna o en base a las enfermedades con las que interactúan. Para la construcción de estas matrices se utiliza:
Con estructura química y secuencias de aminoácidos:
- Algoritmo SIMCOMP.
- Algoritmo Smith-Waterman.
Con relaciones de enfermedades:
- Métricas de similitud y distancia: Jaccard, coseno, distancia Euclídea, etc.
En nuestro caso, escogemos las matrices de similitud de Jaccard, partiendo de los datos de relaciones fármaco-enfermedad y proteína-enfermedad. El resultado tendrá un formato parecido al siguiente, donde los colores más oscuros representan mayor similitud entre pares:
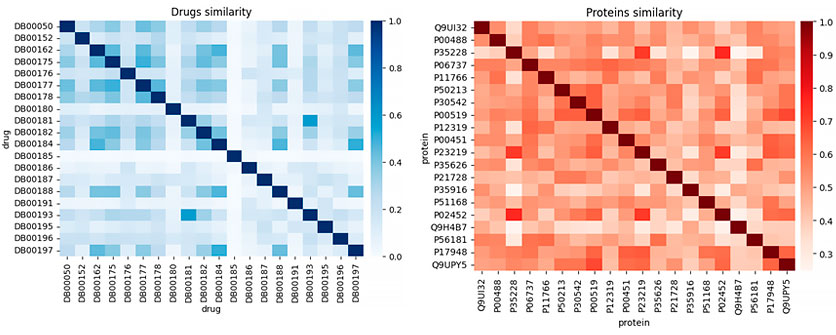
Estas matrices, de una forma natural, pueden ser convertidas en grafos, donde el peso de cada enlace será la similitud entre cada par de elementos. Por conveniencia, se omitirán los enlaces con similitud 0 y los bucles (aquellos enlaces que salen y entran en el mismo nodo).

Vectorización de grafos y base de datos para aprendizaje automático
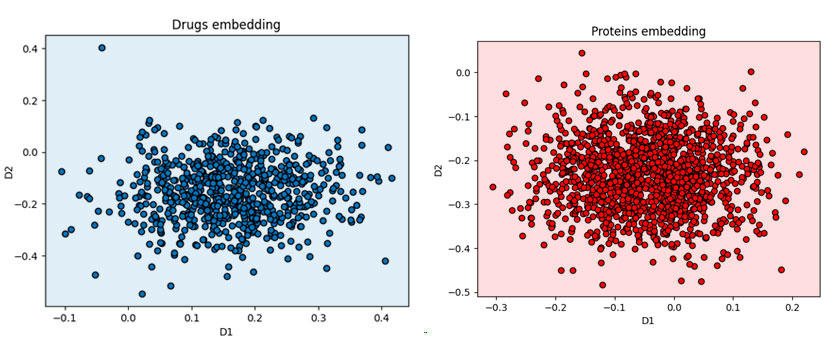
Para poder utilizar la información de los grafos en un algoritmo de aprendizaje automático se pueden convertir a vectores, que son elementos más manejables para los modelos. La vectorización de grafos consiste en transformar cada nodo de un grafo en un vector en un espacio de características de alta dimensión, conservando ciertas propiedades de la estructura original. En este caso, se utilizarán técnicas de caminos aleatorios en el grafo para generar embeddings de características, teniendo en cuenta los pesos de los enlaces para las transiciones. Se elegirá un espacio de 32 dimensiones para representar los vectores.
Un ejemplo de visualización en dos dimensiones de los embeddings de los grafos de fármacos y proteínas es el siguiente:
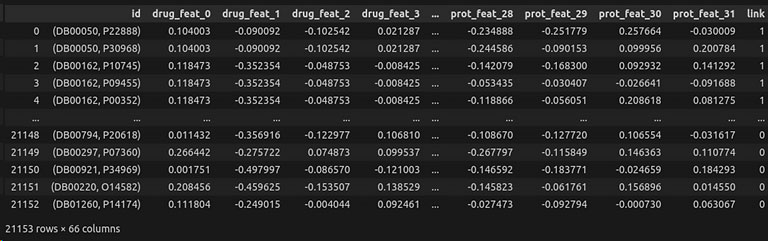
Una vez obtenidas las representaciones vectoriales de los grafos, ya solo queda juntar toda la información para construir la base de datos final que utilizará el modelo de aprendizaje automático. Para crear las etiquetas, se utilizará la información de las interacciones confirmadas y no confirmadas entre cada par fármaco-proteína, siendo 0 si no existe interacción conocida y 1 si existe. Por tanto, cada fila de la base de datos será la concatenación de los vectores de cada par fármaco-proteína y la etiqueta de interacción. Por último, ya que la matriz de links tiene un gran desbalanceo a favor de los negativos, se opta por coger una submuestra aleatoria de éstos para el entrenamiento.
Resultados sobre el reposicionamiento de fármacos
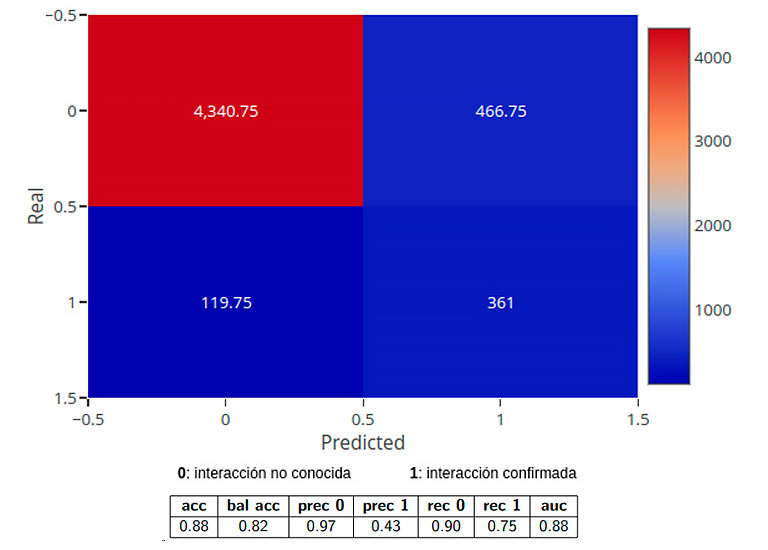
Tras realizar un entrenamiento y evaluación del modelo de aprendizaje automático, las matrices de confusión medias y las métricas se muestran a continuación. Los resultados son satisfactorios, pues el modelo muestra la capacidad de detectar el 75% de los enlaces positivos. Sin embargo, en realidad estos resultados solo sirven para dar una intuición de si el algoritmo está aprendiendo patrones o no, ya que lo importante es fijarse en los falsos positivos (cuadrante superior derecho en la matriz de confusión), que son casos donde el algoritmo ha encontrado probabilidad alta de interacción, pero no está confirmada y serían casos a investigar. De hecho, los falsos positivos están relacionados con la baja precisión positiva (prec 1), lo que indica que hay muchos casos donde el modelo ha detectado un posible enlace no confirmado.
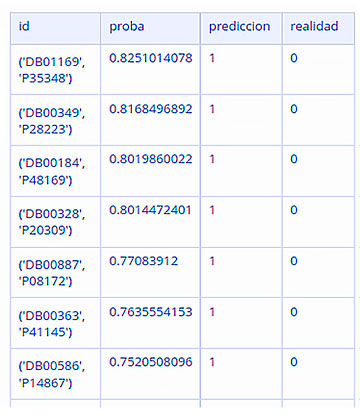
Ordenando los casos de falsos positivos por mayor probabilidad se obtiene una tabla como la siguiente. Estos pares fármaco-proteína (columna “id”) son los que deberían ser estudiados por los investigadores, porque representan potenciales nuevas interacciones.
 Con este estudio, vemos cómo con información sobre fármacos, proteínas y enfermedades se pueden utilizar algoritmos de aprendizaje automático para predecir nuevas posibles interacciones fármaco-proteína. Esto es de gran ayuda para los investigadores a la hora de comprender la forma de actuación de los medicamentos.
Con este estudio, vemos cómo con información sobre fármacos, proteínas y enfermedades se pueden utilizar algoritmos de aprendizaje automático para predecir nuevas posibles interacciones fármaco-proteína. Esto es de gran ayuda para los investigadores a la hora de comprender la forma de actuación de los medicamentos.
En este post hemos utilizado bases de datos públicas sobre fármacos, proteínas y enfermedades. Sin embargo, gran parte de las interacciones negativas son en realidad interacciones no confirmadas, por lo que utilizando bases de datos con interacciones negativas confirmadas (más costosas de obtener) se podría mejorar el algoritmo y que aprenda patrones partiendo de información más exacta.
Desde el área de Salud del Instituto de Ingeniería del Conocimiento (IIC), intentamos aportar a líneas de investigación del sector donde la inteligencia artificial pueda tener cabida. Es el caso del reposicionamiento de fármacos, un ámbito muy prometedor y con grandes ventajas en el desarrollo de medicamentos.